コメントが100件を超えたので、いつものように学とみ子の発言を拝借して、新規の記事です。
ため息ブログは、学とみ子を構わない方が良いです。直接、声かけなどはせずに、遠巻きに、学とみ子デタラメを叫び合っていた方がぼろを出さないで済みます。
ため息ブログが、何かしゃべれば、デタラメ繰り返しが全開になりますから、もう潮時です。
つまり、当方等からのデタラメという批判には反論できないから、勘弁してちょうだいという「泣き」が入ったんですな。
コメントが100件を超えたので、いつものように学とみ子の発言を拝借して、新規の記事です。
ため息ブログは、学とみ子を構わない方が良いです。直接、声かけなどはせずに、遠巻きに、学とみ子デタラメを叫び合っていた方がぼろを出さないで済みます。
ため息ブログが、何かしゃべれば、デタラメ繰り返しが全開になりますから、もう潮時です。
つまり、当方等からのデタラメという批判には反論できないから、勘弁してちょうだいという「泣き」が入ったんですな。
キメラ子にAcr/CAG-GFP という挿入遺伝子と染色体の欠失の有無を調べ、キメラ子はES細胞、今回はFES1由来であるという証明をするため、桂調査委員会はPCRとその産物を電気泳動で調べたわけです。
その調べ方について学とみ子はほとんど理解できておらず、無駄口与太郎も間違えていることを、当方やplus99%さんが指摘してきたところです。
当方の解説を読んで学とみ子は新しい記事を立ち上げました。当方のブログも記事のコメントが100件に近づいたので、この新しい記事を立ち上げることにしました。
いつものように学とみ子は当方やplus99%さんの解説を読んで、そんなことは前から知っているとばかりに書くわけです。嘘つきですから、当方等から情報を得たとは絶対いわないのです。しかしまだ理解できてないから間違いがあるわけですね。
「ため息ブログは、学とみ子の間違いさがしに血眼になっています。」 ← 違いますな。何回も言いますがかっぱえびせんなんですな。学とみ子は当方等が別にお願いすることもなく、かっぱえびせんを供給してくれているのです。
「PCR反応による増幅はどのような仕組みか?について、ため息ブログは、その仕組みを知りませんでした。」 ← ちがいますね。知らなかったのは学とみ子の方です。当方等の説明を読んで理解できてきたのです。その証拠に今回の学とみ子の記事の11月7日(火)午後13時現在の記事の最後は当方のコメントの転載で、この当方のコメントに対しての反論は一つもありません。せいぜい「繰り返すことによりforward primer と reverse primermの間のDNA鎖が指数関数的に増えるわけです。」と書いていますが、こんなことは誰でもすぐに理解できますから、無駄な説明です。特に、下線部分の100bpにこだわる記載は不要です。」 くらいしか書けないのです。
「plusさんは、プライマーではさむという仕組みが理解できず、20塩基あれば、バラバラになっている遺伝子構造の特定部位を選ぶことができるのだから、それだけで十分なんだという極論になってしました。」 ←学とみ子はプライマーの設計が理解できていないからplus99%さんの塩基が20ケ位の塩基配列があれば、全DNA鎖の中から特定の部位を特定できるということがわからないようです。
「端に赤あるいは青部分が付いたDNA配列の構造体となれば、ポリメラーゼは、赤、あるいは青構造を感知して、そこで伸長反応をやめます。」 ← 理解できてない証拠です。赤、青の構造体とはなんだかわかりませんが、そんなのはありません。鋳型となるDNA鎖はその前のステップでの合成開始を示すプライマーなので、これ以上鋳型になる部分がないので合成が止まるのです。多分、この説明では学とみ子にはわからないでしょうから、もっと勉強して理解できたら、この当方の表現の意味が理解できるでしょう。
追記:ため息は親切だから尊大なおマヌケにもわかるように説明知てあげました(下記)。
「ここの理解がやや難しいので、ため息ブログは理解できなかったのです。
他にもいろいろPCR反応説明図はありますが、この図が、「なぜ、挟めた部分のみが増幅するのか?」の理屈を良く説明できています。」 ← お笑いですな。どんな説明にも赤青の構造体があるから合成が停止するなどという説明はありません。
「しかし、プライマー構造も含みはさまれた部分が、なぜ選択的に増幅していくかをしらない人は多かったです。」 ← 学とみ子だけが知らなかったのです。皆さん知っています。
「端にプライマー部分があれば、伸長反応は、その構造を感知して止まるという理屈を、彼らは知らなかったのです。」 ← 完全に娯解してますね。それ以上鋳型になる部分がないから止まるのです。
「長いDNA配列と、短いDNA配列がなぜ、同じように見えるのか」 ← 学とみ子はまだ欠失部そのものが増幅されるから長い配列があると思っているようです。繰り返し説明したのにわからないようですな。
一方、こっちのバカが喚いています。
無駄口与太郎曰く:アルツハイマー野郎が、だったらどうしてTCR再構成バンドはラダーになるのだ。長さが違うからだろうがよ。お前はもう引退せい。社会の害悪だ。
PCR結果を電気泳動で示すという理屈が全く理解できてないのですな。複数の長さの異なるDNA鎖があるとき、これを電気泳動すると、塩基の長さにしたがってDNA鎖は移動するのです。その結果電気泳動の図には複数のバンドが出現し、梯子=ラダーというわけですな。PCRの結果は、特定の長さのDNA鎖ができるわけですから、バンドは一つだけです。あるいはPCRで増幅できる鋳型がなかったのでバンドはなしです。All or Nothing なのです。これがわからないのですかね。理解できていないのに罵倒するから、バカとしか評価されないのですな。
クリックすると拡大されます。
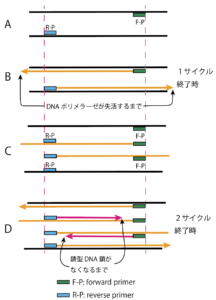
黒い線がオリジナルのDNA鎖です。F-Pはforward primer、R-Pは reverse primer です。DNA鎖には方向性があって3’、5’と表記しますが煩わしいので省いています。追記:学とみ子が理解できてないので改訂しました。プライマーが鋳型DNAに結合するのと、合成されたDNA鎖の一部になっている違いも示しています。
加熱で乖離したDNA鎖に設計したforward primerとreverse primerが付きます(A)。DNAポリメラーゼがそれそれ矢印の方向へDNA鎖を合成します(B)。合成が終了するのは温度を下げてDNAポリメラーゼが失活するまでです。オレンジのDNA鎖ができたわけです。ここで加熱するとforward primerとreverse primerが再び結合します。結合する部位はオリジナルのDNA鎖では前と同じで、オレンジのDNA鎖には図のような部位に結合します(C)。DNAポリメラーゼが働くように温度を上げると、オレンジのDNA鎖は1回目と同様に合成されていきますが、これに加えてオレンジのDNA鎖が鋳型になりピンク矢印のような方向に合成が始まります。このピンクのDNA鎖の合成はオレンジのDNA鎖が合成を開始したときにできた部分(プライマー)まで進みますが、それから先は鋳型になるDNA鎖がないのでここで停止します(2サイクル目の終了:D)。結果ピンクの長さのDNA鎖ができます。この合成が停止するのはなにか停止させる構造体があるからではないのです。鋳型がないからです。3サイクル以降では、2サイクル目同様オレンジを鋳型とするのに加え、ピンクのDNA鎖を鋳型にしてピンクのDNA鎖がつくられるわけです。したがってnサイクルを繰り返すと、オリジナルにの黒いDNA鎖は2本のままかわらず、オレンジのDNA鎖(2n-2)本でき、ピンクのDNA鎖は(2^n-2n)本できることになります。ピンクのDNA鎖が指数関数的に増加していきます。
はい、学とみ子様おわかりでしょうか?そんなのはわかっているなどと決して言わないでください。「赤、あるいは青構造を感知して、そこで伸長反応をやめます。」と言ったのですからね。理解できたのなら、おかげさまで理解できたと言うのですよ。社会人の常識ですからね。
動画の1分〜 を見てみな。