審査書類情報照会の特許出願番号に 2017-501108 を入れて 「照会」 ボタンをクリックする。
2. 2016/11/11 :国際出願翻訳文提出書
—-/–/– :明細書
—-/–/– :配列表
—-/–/– :請求の範囲
—-/–/– :要約書
—-/–/– :図面
が出てくるから。
明細書 をクリックする。【図面の簡単な説明】【0006】に、
「 【図20】SACおよびCD45+細胞由来のSACから誘導されたキメラマウスのTCRβ鎖再編成解析を示す図である。2Nキメラマウス#1、#2、#3、#5、#6、#7、#8および#9に再編成DNAの発現がみられた。」とある。
SAC とは明細書の【0180】に「ストレス変化幹細胞(Stress Altered Stem Cel(SASCまたはSAC)と呼ぶ。」とあるから酸浴して生き延びた凝集した細胞群=STAP細胞から作成した幹細胞=STAP幹細胞のことでしょうね。
図面をクリックし、図20をスクロールして探す、
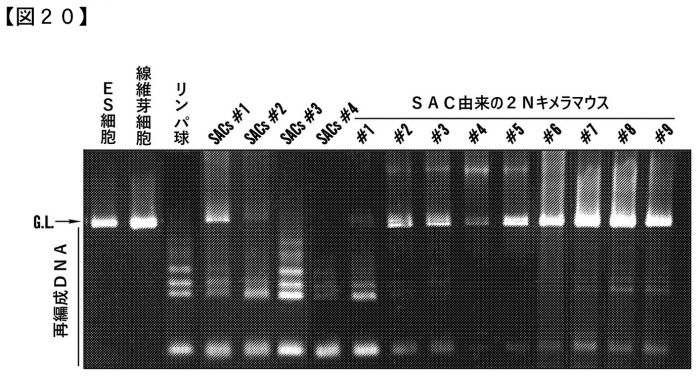
STAP細胞由来のキメラ動物#1〜#9でTCR再構成があったことを示している。この記事のタイトルに再編成とあるのは申請書にあるからですよ。
学とみ子さん、何故この図が論文にないのでしょ?
[ 追伸 ] 2018.11.30 10:05
2018/11/30(金) 午前 8:24 学とみ子 に
勉強させてもらってます。この先、当該記事を消さないようくれぐれももお願いしたいと思います。
とのご返事をいただきました。このあとの応答に期待します。しかし、消すなとはどういうことだろ?
この特許申請書では申請者から小保方ははずされていますね。なぜでしょう。
しかも、理研で不正認定され、筆頭緒著者も捏造を認めたArtcle Fig.5c と審査書類の図24Bは酷似しています。
信用できない特許申請書類ですね。
学様へコメントしました。
学様曰く
>STAP論文では、Phに幅がありますね。最初は、職人芸でしょう。だんだん、結果が安定してきて、実験条件が固まって来ると思います。
だそうです。そんな状態で論文を出しても意味がないでしょう。結局第三者による再現ができずに消えていく論文になるだけですがな。
本当に論文を書いたことがあるのですか?
もう笑うしかないです。
山の住人さんのArticle Fig.5c は“細胞増殖度グラフ”の方ですね。 Fig.5c も研究不正認定されていますが、Article Fig.1i が、研究不正認定された TCR 遺伝子再構成図。
TCR 遺伝子再構成を示すデータ・図は、以下の3つあります(表に出ているもの)
(1) STAP 論文 Article Fig.1i
(2) 国際特許出願 WO2013/163296 A1 Fig.20(本ブログ記事の図と同じもの)
(3) 調査委員会に“小保方氏の TCR 遺伝子再構成の実験結果(PCR)”として提出されたもの(例のGel1 & 2)
上記の(1)と(2)は 2N キメラマウスによるデータの有無に違いがあるものの、(3)のデータによるものであることは明らかなので、問題となっている TCR 遺伝子再構成の実験者は“小保方氏”であることも分かるわけで、学とみ子氏はそれにも関わらず、小保方氏ではなく、他の若山研メンバーによるものだと吹聴していますね。
ため息先生の本ブログ記事に於ける『学とみ子さん、何故この図が論文にないのでしょ?』という質問・疑問に対し、学とみ子氏が答えると思いませんので、私が答えましょうか…
まず、TCR 遺伝子再構成のデータの問題には大きく分けて 2 つあります。
〈1〉Lymphocytes の部分の切り貼り(研究不正認定されたもの)
〈2〉TCR 遺伝子再構成に関する不整合データ隠蔽疑惑
〈2〉の詳細は、調査委員会による報告書p27-28 に記載されているもので、不整合データ隠蔽は研究不正のうちの“改ざん”にあたりますが、委員会は共著者の“その経緯の説明”から『意図的な隠蔽ではなく、研究不正とは認められない』と判断されています。
次に小保方氏のデータがどのように扱われたかを考えなければなりません。
分かっているだけでも、それは 3 つ考えられます。[1] 小保方氏から若山研でプログレスレポート等で共有されたもの、[2] 小保方氏が笹井・丹羽両先生に論文を書く段階で共有されたもの、[3] 小保方氏が直接、バカンティ氏側に共有したもの、の 3 つです。
[1] と [2] に関しては、論文作成側つまり理研側の問題なので“〈2〉TCR 遺伝子再構成に関する不整合データ隠蔽疑惑”の詳細説明から分かるように、ある一定の科学的理由のもとにデータを出さなかったということになっています(出さないと決めたのであれば、TCR 遺伝子再構成のデータを中途半端な出し方、『調べた』と書いておきながら、それに対するデータが無いとか…色々問題はありますが…)
しかし、“[3] 小保方氏が直接、バカンティ氏側に共有したもの”に関しては、論文作成側つまり理研側の問題ではなく、バカンティ氏側の知財戦略上の判断があります。ここには科学的判断とは別の判断があります。
日本人の考える知財戦略と海外の人の考える知財戦略はその国々の特許制度からくる判断が異なります。特にアメリカの場合、怪しかろうが出鱈目であろうが、科学的理由にキチンと裏付けがなされていなく疑わしくても、取り敢えずデータを出すなんてことが数多くあります(以前、STAP 特許で特許詐欺になるのか、という件でコメントしたことがあります。)
実験的に科学的裏付けがなされていなくても、理論的にあり得ると特許審査官が判断した場合、特許がおりる場合があります。
しかし、STAP 特許はその元になった論文、データに既にネガティブな見解が数多く出ていますので、審査官がその見解を判断材料に入れないはずも無いので、拒絶査定そして特許範囲の分割・減縮の繰り返しになります。今、『酸による遺伝子発現の亢進等』にまで縮減されているのですかね? それ自体は新しくとも何とも無く、“新規性”が認められ無いですよ。
あちらの学とみ子氏のブログでは、もう一つの STAP 特許(ヴァカンティプロトコル版?)についても、話題にしているようですが…
STAP 特許関連はもう日本の中での問題、小保方氏の問題では無いですよ。
(調べましょうかね…もう面倒くさいので嫌なんですけど(´-ω-`))
—–
plus99%
hidetarouさん
>ヴァカンティ教授と小島医師が発明人のもう一つのスタップ関連の特許
それは私のコメントに対するもの?
特許に関しては詳しい人に聞いた方がいいと思いますけど、その特許が認められると小保方氏にリターンがあるとお考えで?発明者が別の方になるようですが。
前の特許でも早期に関係者ではなくなっていたようではありますが。
(略)
2018/11/28(水) 午前 11:09
—–
hidetarou
もう一つのスタップ関連の特許では実施例でスタップ幹細胞が使われている様ですが、特許に詳しい人の意見を聴きたです。
2018/11/28(水) 午後 8:23
—–
oTakeさん
>STAP 特許関連はもう日本の中での問題、小保方氏の問題では無いですよ
これが意味がふた通りに読めるのですが。
バカンティ氏の特許はすでに、日本とも関係ない、小保方氏とも関係ないという意味なら私はそういう認識です。
バカンティ氏は不正に関与したという認定はされていないので、STAP論文からでも使える材料はなんでも使うでしょうし、174だかある請求項も数撃ちゃあたる式なのだと理解していますが。
STAPという名称のものを出願に含めるのは出資者に対するポーズとして意味があるのでしょうが、発明者に含めなかったことが象徴的だと考えています。
oTakeさんが貴重なお時間を使っても、擁護の方々は都合のよいところだけつまみ食いするだけでは。
このゲル写真は、Scienceのfigureでも使われたものでしょうか? もしそうなら、Scienceのreviewで ”spliced in” と指摘されている ”lanes 3 and 6”(別スレでの体内時計さんのコメント参照)というのは、どのレーンなのでしょうね? Lane 3は、このゲルでも3番目の「リンパ球」とラベルされているレーン(切り張り確定済み)である可能性が高いと思われますが、lane 6はどれでしょう? 順に追えば、6番目のSACs #3かと思いますが、このレーン(と次のレーン)はGLのバンドが非常に弱く、CD3で純化した細胞からのSACでないと説明つかないかもしれませんね。
個人的には、 SAC由来2Nキメラサンプルの#1が興味を引きます。これが本当にtail tipのサンプルだとすると、血球混入では説明できないレベルのTCR rearrangeが見られる事になります。T細胞由来STAPがキメラに貢献した証拠になり得るので、このキメラは徹底的に解析すべきだったと思うのですが、、、
一方で、Scienceのreviewで指摘されたlane 6が、キメラ#1のレーンだった場合は、意図的な切り張りの可能性も否定できません。その場合は、lane 3の「リンパ球」レーンの切り張り(これは科学的結論にほとんど影響しない)より、はるかに深刻な問題になり得ますね。
この特許のゲル写真の切り張りについて、 詳細ご存知の方がおられたら、教えてもらいたいです。
>L さん
『このゲル写真は、Scienceのfigureでも使われたものでしょうか?』ですが、Science 誌の Review の文字情報として、流出していましたので、分かりますが、実際の TCR 遺伝子再構成図については、Science 誌側と小保方氏(ヴァカンティ氏は知っているかもしれません)しか知らないはず…です。若山先生も図は見ていなかったはずです。調査委員会にも提出されていなかったと思います。
Nature 誌の TCR 遺伝子再構成の図はlaneが 5 つしか有りませんが、特許出願のFig.20の lane 1 〜 5 が Nature 論文のFig.1i のものと一致しています。調査委員会がこのFig.1i の元図(Gel1 & 2)を手に入れていますが、特許出願図 Fig. 20 と Gel2 の 2N キメラの部分が一致していないようです…元図 Gel2 に 2N キメラマウスのものがありますが、括弧書きで CD45 STAP と記載があります。“Lymphocytes”が“CD45+/CD3+ 1(100 ng) ということは、対応する 2N chimera 1 〜 9 (CD45+/CD3+) とラベリングされたものが別にあるということになります。元図 には Gel3 なるもの(非公開)が存在している可能性がありますね。
Fig.1i の元図(Gel1 & 2)は画像加工された痕跡が無く、元図(Gel1 & 2)のものが用いられています。特許出願図 Fig. 20 は確実に恐らく Gel3 等他の画像からの切り貼り合成等の加工がある可能性が高いです。
《物凄く気になる点》
調査委員会が手に入れた元図(Gel1 & 2)に “STAP cluster(STAP 細胞塊)”と付いた lane があります。lane 5 & 6 には“Sorted Oct4+”と記載されています。ヴァカンティ氏はSTAP cells の意で“Stress Altered Stem Cell (SASCs or SACs)”も使われています。
“Sorted Oct4+”と“SACs”と“STAP cluster(STAP 細胞塊)”とどのように違うのか…さらにはヴァカンティ氏の特許宣誓書には“stress-produced stem cell (SACs)”とあるようです…
また、小保方氏は(Animal )Callus(STAP 細胞)の Callus1 と Callus2 を STAP 細胞(Callus1) と 元細胞のCD45+ 細胞(Callus2)に書き換える(調査委員会報告書p26)なども行なっています。
STAP 細胞塊に対し、4 つの呼び名があり、その中には元細胞に変えたものがあったりしています。ラベリングそのものを信用するかと言ったならば…信用出来ないですよね…コレは…
色々ありますね…不可解な点が…
ザクッとしたコメントですが、参考になりましたでしょうか?(混乱するだけかもしれませんが…)
訂正)
Fig. 20 は確実に恐らく Gel3 等他の画像からの切り貼り合成等の加工がある可能性が高いです。
→Fig. 20 は Gel3 等他の画像からの切り貼り合成等の加工がある可能性が高いです。
https://blogs.yahoo.co.jp/solid_1069/15765637.html
より、
>学とみ子はアクセス数もさほど多いわけではなく、影響力も少ないでしょうけど、アンチSTAPの方々には何らかの脅威なのでしょうか?
とありました。
2年前なら
https://blogs.yahoo.co.jp/solid_1069/15759910.html#15765957
のようなコメントが投稿されると、「特許申請されている→STAPは本物→擁護派歓喜の雄たけび!」になっちゃうところだったわけです。
このようなことを再発させないためにも、デマは徹底的にたたく必要があるのです。
件のブログ主様は、自らのブログの影響力を軽視しない方がよろしいかと。
【未確定情報にて要注意】
(1) “CD45+/CD3+2(100ng)”を“SACs#3”とすると、“Lymphocytes”を STAP 細胞としていることになりますね。
(2) 特許出願図の Fig20 のSACs#4 〜 SAC由来 2N chimera#5 が別のGelか?
Gel3?
(3) SAC 由来 2N chimera#6 〜 9 は Gel1 の11 〜 14(STAP cluster 1 〜 4) とすると、これは 2N キメラマウスでは無い可能性が高くなりますね。
(確定するには鮮明な特許出願図Fig20が必要かと…)
—–某掲示板による情報…
小保方氏が提出したゲルを見ても、明らかにSACs#2の隣はSACs#3ではない。 小保方氏が提出したゲル2枚を拡大して見ると、特許図20のSACs#3はGel 2の18番(CD45+/CD3+2(100ng) 特許図20のSAC由来の2Nキメラマウス#6〜#9はGel 1の11〜14(STAP cluter1〜4) SACs#4〜SAC由来2Nキメラ#5はバックグラウンドの明度が違うから別のゲルからの切り貼りか、査読がAlso I find the leading edge of the GL band suspiciously sharp in #2-#5. と指摘しているから、合成になっているのかな?
—–
L さん oTake さん

この特許申請の図は解像度も悪くダウンロードして拡大してもはっきりしません。モノクロの閾値を変えてもはっきりした切り貼りはわかりにくいです。
oTake さんがおっしゃるように。この特許申請の図20のレーン1〜5はリンパ球のレーンが切り貼りしたことが確定したNature Article Fig.1i ですので、この特許の図20をこのことを踏まえて調べてみました。Phooshopでダウンロードしたjpgの図のレーン1〜7の部分の明るさを+60%、コントラストを-30%にしてみたのが下の図です。
レーン3(リンパ球)が両側と異なる背景なので認定された通り切り貼りがわかります。レーン6(SACs#3)の上の方にレーン5(SACs#2)と背景が直線的に区切れており、レーン6の下の方はレーン7(SACs#4)と背景が異なることがわかります。なおかつ、レーン6と7の背景n違いが右肩上がりの斜めです。レーンが左に傾いているのをコピペしたのではないでしょうか。とするとGel2 で上が左に傾いているのは16〜21番レーンでその中で18番レーン(CD45+/CD3+2(100ng)ng)が一番似ている。
Scienceのレフリーにレーン3と6が切り貼りだと指摘されていることと一致しているように思えます。
学とみ子さんは頻繁においでになっているので、この記事、コメントを読んでいると思われ、問いかけもあるのですが、反応がないので、「“STAPあるべき論”の...」 にコメントを送って見ました。2018.11.29. 10:19頃。そのコピーです。
TCR再興性 → TCR再構成 変換ミスです。かのブログにも修正コメントしました。3018.11.30 10:10
ため息さん、私がこれまでに見た記憶があるのは、oTakeさんのおっしゃるGel 1&2だけで、今回のは初めて見ました。色々な意味でちょっと驚きです。特許に疎いので、基本的な事で申し訳ないのですが、何点かお伺いしてよろしいでしょうか?
(1) この写真は、2016年11月16日出願の国際特許での図、すなわち小保方さん抜きでVacantiが英語で出願した書類の翻訳版に含まれている図、という理解でよろしいでしょうか?
(2) その場合、この写真はVacantiが自分で特許書類に新たに加えた図と考えてよろしいでしょうか?
もしそうであれば、この写真はVacantiの手元にある何らかの図表をコピペしたものではないかと考えます。STAPがらみで過去に論文投稿されたバージョンはすべて、Vacantiに送られていると思いますので、そこで使われた図表からのコピペではないかと邪推しています。最後のNature版にはこの図は入っていないので、Science版あるいはそれ以前の投稿で用いられた図を引用し、ラベルだけ翻訳された可能性を考えます。
荒い画像にもかかわらず、解析どうもありがとうございます。素直にSACs#3をlane 6の切り張りと捉えるのが妥当なようですね。とすると、SAC由来2Nキメラサンプルの#1が、一体何のサンプルなのか、色々な意味で興味深いです。
oTakeさん、色々とありがとうございます。図の切り張りのストラテジーについては、何となく理解できた気がします。いずれにせよ、SAC由来2Nキメラサンプルの#1が、どのような由来なのか、情報がないと何とも言えないですね。
個人的には、SACとSTAP clusterは同義(細胞塊をそのままピックアップしてゲノムDNA抽出したサンプル)と考えています。“Sorted Oct4+”は、光っている細胞塊を集めて、一回シングルセルにしたのち、FACSでGFP高発現細胞をソートしてゲノムDNA抽出したサンプルと思われます。ソートがきちんと行われていれば、“Sorted Oct4+”サンプルは、初期化された細胞が高純度で集められたものと解釈可能ですが、STAPチームはFACSをきちんと使えてなかった可能性が高いので、ソートの意味がどれほどあったかは不明です。
L さん
当方も特許申請については全くのド素人ですので、発明者にバカンティ氏と小島氏しかいない、なぜだろ?位しかわかりません。
生データは一括して小保方氏が管理している(できているかどうかは別にして)ように思えるので、おっしゃるように、バカンティ氏の手元にある図・写真は投稿寸前のコピーだと思えます。最終投稿原稿に使わなかった図も、論文のリバイスに伴い持っている可能性があります。この特許申請のために、別途図を小保方氏に依頼したとは、思えないですね。もしそうなら、小保方氏も名前を連ねるべきで、そうではないところを見ると、バカンティ氏は手元にある図・写真で勝手に特許申請したと考えるのが妥当かと推測できますが、事実は、バカンティ氏は墓まで持ってい行っちゃうようで、わかりませんね。
それぞれの図・写真の説明ができるのは小保方氏だけなんでしょうね。御本人も記録がないからできなくなっちゃっているのかもしれない。
5ちゃん情報ですが
0471 名無しゲノムのクローンさん 2018/11/29 13:38:00
今話題の特許図20は
米仮特許出願(2013.3.13) US61/779,533
から登場し(Fig.21)
国際特許出願(2013/4/24)PCT/US2013/037996
にも載せられている(Fig.20)
だそうです。
>L さん
(1) この写真は、2016年11月16日出願の国際特許での図、すなわち小保方さん抜きでVacantiが英語で出願した書類の翻訳版に含まれている図、という理解でよろしいでしょうか?
(2) その場合、この写真はVacantiが自分で特許書類に新たに加えた図と考えてよろしいでしょうか?
2016 年 11 月 16 日出願の国際特許での図はあくまでも翻訳版の公開文書です。
実際には 2013 年 3、4 月に国際特許としてデータが提出されたものが元になっています。Nature 誌での論文が出る前です。
この文書は、2013 年 10 月 31 日に “Application Body as Filed” としてWIPO(世界知的所有権機関)から公開されています。
《経緯》
2012 年 3 月に西川先生の TCR 遺伝子再構成のアイデアを受け、小保方氏はその実験を行ない、若山研で TCR 遺伝子再構成があったとプログレスレポート等で報告(時期は2012年中頃で以下のリジェクト 2つの前か後かは不明)
2012 年 6 月 6 日に Cell 誌へ投稿し不採択となった原稿には切り貼り TCR 遺伝子再構成データが含まれており、この一部が 2013 年 3 月 10 日投稿の Nature 誌での切り貼りされた TCR 遺伝子再構成のデータとして使われている(理研CDB の調査結果より)
Science 誌は 2012 年 8 月 21 日に「我々の限られた誌面では、論文を修正しても掲載できない」として、事実上のリジェクト。このリジェクトに至るまでの Reviewer コメントにも切り貼り等の問題の指摘があります。これを元に小保方氏が 2012 年 12 月 11 日 版として改定。笹井先生が論文の更なる改定をし、Nature 誌のベースとなる論文が 12 月 28 日に完成。2013 年 1 月上旬に小保方氏はこのベースとなった論文をどうするかを相談する為、ブリガムのヴァカンティ氏のもとを直接訪れています。
同時に、2013 年 1 月に丹羽先生が論文作成に加わり、その際、TCR 遺伝子再構成に関して論文に含めるかどうかの話になっています。恐らく、この時点で国際特許図 Fig20 と Nature 論文での Fig.1i の差、STAP 幹細胞・キメラマウスの有無が明確に分かれたのだと推測します。
2013 年 1 月頃から、小保方氏は主に若山研ではなく、笹井先生の研究室にいたことが分かっています。この間に Letter 論文を作っています。
そして、Nature 論文 2 報の初稿は 2013 年 3 月 10 日。
(因みに、2013 年 4 月(4日?)にScience 誌のリジェクト理由等、Nature 誌側は切り貼りの可能性も含めて全部把握していたようです。)
その後、2013 年 4 月 24 日、USPTO に国際出願。この出願書類には、論文からのデータが追加。この特許は、2013 年 10 月 31 日に公開された(特許書類 WO 2013/163296)。これが上記の“2013 年 10 月 31 日に “Application Body as Filed” としてWIPO(世界知的所有権機関)から公開されています。”にあたるものです。
以下の WIPO のページから見ることが出来ます。該当の文書は下から5番目です。
https://patentscope2.wipo.int/search/en/detail.jsf;jsessionid=670FF85560828652201CBE3383B7711E?docId=WO2013163296&tab=PCTDOCUMENTS&maxRec=1000
話題の特許は、こちらが大元です。
https://bit.ly/2Rt1AK8
2014年3月19日出願の2件と2014年8月28日出願の1件に対し、優先権を主張して1件の出願にまとめて、2015年9月24日に公開(Publication Date)となっているようです。
こちらのFIG.20も、あまり解像度は良くないです。
軒下管理人さん
モノクロ2階調に変換するとき目の荒いハーフトーンとかにした図を使い回しているようですね。元の撮影写真とかScienceに投稿したpdfでないとわからないですね。
24.09.2015 Priority Document の p33 にも Fig20 が使用されていますね。
WO2013163296:GENERATING PLURIPOTENT CELLS DE NOVO
こちらは“inventer(発明者)”は、フルメンバーです。
WO2015143125:METHODS RELATING TO PLURIPOTENT CELLS
こちらは“inventer(発明者)”は、ヴァカンティ氏と小島氏のみです。
元画像が欲しいところですね。
今、比較的画像分析出来るものとしては、Gel1 & 2 ですね。
逆転の発想で、特許図 Fig20 からハーフトーンのパターンを割り出して、Gel1 & 2 の画像を、同様にハーフトーンにして、比較する…元図から“回転”や“部分的改変”があったら面倒くさいですが、何か分かるかも!
因みにそんな面倒くさい解析を誰がそれをやるのか(´-ω-`)
oTakeさん
そうですよね。キメラでTCR再構成があったのなら堂々と図示すべきですけど、特許申請にはあって、論文にないのは何故でしょね。丹羽氏はキメラのTCRを否定しているのだから、このGel1、Gel2 どこまで信用できるのかから始まって、これから合成された特許申請の図20はナンジャラホイですからね。
もう撤回された論文の結果を、あそこが捏造だ、いやちがうなんて議論する意義はないですからね。
でも、中毒になってしまったので、有る限り止められない…トホホ。
sighさん
特許申請は、日本では電子ファイルでも可能です。アメリカでも同様でしょう。
写真は、電子ファイルにする時に、ファイルサイズを大きくしなくて済むように、粗い状態で保存することになっていると思います。
部品の断面図などでも、ハッチングの種類を変えることはあっても、写実的な濃淡は付けてはいけないようです。
ということから、特許のFIGから、オリジナルの写真の状態は、分からないです。
>元の撮影写真とかScienceに投稿したpdfでないとわからないですね。
そういうことです。
トホホなかーま(笑)。
ここまで来ましたので、トコトンお付き合いさせていただきます。
改めて思うのですが、STAPを擁護する方々は、何が面白くてこのような過去の遺物を発掘するのでしょうか?
この特許も、件のブログに投稿されなければ、我々の目に留まることはなかったのに、と思います。
擁護しているつもりで、結果、STAP論文の瑕疵をわざわざ増やして晒しているように感じます。
楽しいのでしょうか?楽しいのでしょうね。私も楽しいです。中毒ですから。
裏を返すと、禁断症状が怖い。
キメラのTCR再構成については、理研の中間報告のスライドにあった2Nキメラの電気泳動図が、その後の修正版では削除されていたことが話題になりましたね。理研は削除した理由を「未発表のデータが含まれていた」と説明したそうですが、釈然としません。
修正前:http://web.archive.org/web/20140314222554/http://www.riken.jp/~/media/riken/pr/topics/2014/20130314_1/document-5.pdf
修正後:http://www.riken.jp/~/media/riken/pr/topics/2014/20130314_1/document-5.pdf
理研の中間報告のデータ、特許申請書のデータについて、詫摩さんがブログに詳しく書かれていました。
こちらの皆様にはわかりきったことだと思いますが、詫摩さんのブログは素人にもわかりやすいので助かります。
吉村先生がコメントされていたので、一部引用させていただきます。
http://blog.miraikan.jst.go.jp/topics/20140317stap-2.html#comment-2609
***********
もとさんの『一つの細胞から分化した細胞ならTCR再構成で見えるバンドは1つ(または2つ?)のはずなので、こんなにいっぱいバンドが見えるのが何故かは私にはさっぱり判りません。』は完全に正しい推論です。胚盤胞に入れられる細胞はせいぜい20個でそのうちマウス組織になるのは数個なので、この方法で見れるバンドはせいぜい1本です。この2Nキメラの解析結果が不自然であることを理研の先生たちは理解しているからこの図をもって『キメラにTCR再構成がある』と言わないのだろうと思います。
***********
体内時計さん
ありがとうございます。石井委員会の改訂前のpptをpdfにしたファイル、魚拓があったのですね。この改訂前のGel2に2NChimeraのレーンが3本あるんですけど、特許の9本のレーンのどれに対応するのか分からないです。
吉村氏の解説によるとTCRのバンドは2あるいは1本ということになると、Gel2、特許の図20ともにおかしいことになりますね。
石井委員会の発表でキメラのレーンが見えないように改訂したのも、突っ込まれて、さらに解析したくなかったからなのかもしれませんね。このときは、理研はなんとか批判をかわしたかったようですからね。
oTakeさん
4 つの呼び名:バカンティ氏は笹井氏の作ったSTAPを使いたくなかったのでは。オリジナルは自分のところにあるんだから、こっちで名前を付けさせろということでは?
oTakeさん
Photoshop でモノクロ2階調に変換は:
入力 300 pixel/inch 出力 300 pixel/inch (要するにサイズを変えない)
ハーフトーンスクリーン
行数 75 角度 45 度 菱形
がもっとも近そうです。あとはGel 1、2 の明るさとコントラストを調節して比べればいい。 Gel 1,2 には結構ゴミがあるのですが、そのゴミが特許申請書 図20にない。
日本の拒絶された、筆頭著者の名前のある申請書にも、日本語のフォントがちがうだけの同じ図があるね(特許情報プラットフォーム 2015-509109 で検索)。こっちは図の説明では「TCRβ鎖再構成分析」というわけで再編成という言葉は使っていない。
【脊髄損傷治療について?】
学とみ子氏ブログにて
—–
hidetarou
学さんへ
ティブレークさんのブログも参考にしてください。
https://blogs.yahoo.co.jp/teabreakt2/18082069.html
2018/11/30(金) 午後 10:55
—–
根本ブログを除いて見た。
以下の JISAI 氏のコメント引用が少し気になりました。
—–
(略)
新実験の方は、成体ラットの脊髄損傷治療として、脊髄ニューロン(痛覚過敏応答と熱応答)の喪失後に脊髄ニューロンのSTAP幹細胞移植で機能回復できたらしいです。
要するにこの新規特許は、2014年9月のSTAP追加プロトコルをベースに特許申請されたもののようです。
2016/1/17(日) 午後 2:06 [ JISAI ]」
—–
脊髄損傷治療について
脊髄の細かい構造の中に、分化済みの細胞を移植することは技術的に不可能。それ故に、未分化な神経幹細胞を移植し、脊髄中で適切に分化、遊走させ、移植先の神経細胞や神経ネットワーク、髄鞘を形成させる(私の知識ではこの程度までですが…)
少なくとも再生医療で多能性幹細胞(ES 細胞、iPS細胞)を使う際、“未分化な神経幹細胞を移植する”となると“神経幹細胞”まで、分化誘導させなければいけないと考えますが…移植医療の場合、それらがそのまま移植されることはないですからね。“脊髄ニューロンのSTAP幹細胞移植”というのは考えられないですね。医療的な“エビデンス”はあるのでしょうか…うーん、かなり怪しい(´-ω-`)
確か、STAP 論文が出て、2014 年にヴァカンティ氏が『脊髄損傷治療で STAP 細胞を移植して、犬が歩けるようなった』とか、しばらくして…『脊髄損傷治療で STAP 細胞を移植して、猿が歩けるようなった』とか…そして、その治療した猿は何処にということになりまして…
『猿には逃げられました(´⊙ω⊙`)』
『ちょーっと待てや、おっさんΣ(-᷅_-᷄๑)』
というお話が…笑っていいのか、悪いのか…
それで霊長類の動物実験で逃亡はヤバイでしょとか言っていた記憶が…
それでお次は『成体ラットの脊髄損傷治療…』(ラットなら大丈夫だと思ったんでしょうか…)
時系列で並べると
2014年3月19日2件出願
2014年3月20日バカンティプロトコル公表(ガラス管に通すことが追記されていると考えられる)
2014年8月28日1件出願
2014年9月3日バカンティ修正プロトコル公表(ATPを加えることが追記されていると考えられる)
優先権を主張して最初の出願の1年後に1件の特許(WO/2015/143125、PCT/US2015/021418)にまとめる。
ということです。
プロトコルを公表する前に特許出願。基本中の基本。実に分かりやすい。
oTakeさん
ド素人の素朴な疑問なんですが、Vacanti氏の主張通りSTAP細胞をそのまま脊髄に移植したら、脊髄腔にテラトーマを作成することになってしまうのではないでしょうか?それに移植する細胞が未分化であればあるほど、細胞が分化する過程でがん化する危険が高いのではないかと思うのですが?
学とみこさんがこの件で記事をアップしました。
いえいえ、そんなことはないですWelcomeですよ。どうぞ、いらしてください。
生データを管理しているのは小保方氏でレーンの切り貼りという捏造したのも小保方氏ですから、切り貼りしたデータに、oTakeさん曰くのGel3とかもあって、加えて作ったのでは?
小保方氏が石井委員会にGel1とGel2の生データを提出し、切り貼りの説明をしたんでしょ。Gel写真を全部提出しないと信用されないからね。そしたらキメラのレーンがあったわけだ。
ゲル写真そのものは有ると思えるけどレーンのラベルが怪しい。STAP細胞からキメラはできないのに、そしてES細胞から作ったキメラだったらTCR再構成のバンドはありえないからね。それでは図20のあるいはGel2のキメラのレーンはナンジャラホイ?何を電気泳動したんでしょ?
桂委員会報告書p28には小保方氏他からの聞き取り調査で、STAP細胞と一部のSTAP幹細胞のTCR再構成実験では最初TCR再構成あったと小保方氏が報告したものの、その後の追試では確認できなかったとある。とすると特許図20の9本のレーンはなんでしょね?
で、学とみ子さん、質問をくりかえしますが、何故キメラにTCRがあったという決定的な証拠になるこの特許の図あるいはGel2が論文にないのは何故でしょ?
アノ姐さん
テラトーマ作成実験ですよね。iPS細胞の臨床試験ではパーキンソン患者さんへドーパミンを作るまで最終分化した細胞を移植するように、iPS細胞を本来のドーパミン細胞を作る黒質に移植するなんてことはやりませんからね。
ため息先生
>石井委員会の発表でキメラのレーンが見えないように改訂したのも、突っ込まれて、さらに解析したくなかったからなのかもしれませんね。このときは、理研はなんとか批判をかわしたかったようですからね。
理研はとにかく“STAP細胞=ES細胞”ということにはしたくなかったのですよね。
研究者の中にも「ESだったら理研は終わりだ」という意見を述べられていた方もいました。
「捏造の科学者」P.178に、小保方氏の会見後、須田さんと丹羽氏が電話で話すシーンがあります。
須田さんが丹羽氏に「STAP細胞由来のキメラマウスの胎児と胎盤」について確認すると、丹羽氏は『隠しても仕方ない。あります』と認められた、とあるのですが、私はこの丹羽氏の『隠しても仕方ない』という言葉が引っかかっています。
この時期、丹羽氏はSTAP細胞の存在を信じていたはずなので、「STAP細胞由来のキメラマウス」についてネガティブに捉える必要はないと思うのですよね。
これは推測ですが、この丹羽氏の発言と、中間報告書で削除したキメラのレーンと、何か関係があるとは考えられないでしょうか。
「捏造の科学者」P.162に、残存試料の解析について、相澤氏の
『STAP幹細胞由来のキメラマウスも残っているが、それを調べても、STAP細胞があったことの証明にはならないから、この検証では調べない』
というコメントが記載されていますが、「STAP細胞由来のキメラマウス」については「調べない」とは仰っていない様です。
「STAP細胞由来のキメラマウス」があるのに解析しないとは考えにくいですし、解析した際、中間報告書で示されたTCRのデータと異なる結果が得られた為、キメラのレーンを削除したのではないか。その様には考えられないでしょうか。
理研は削除の理由を「未発表のデータだったから」としてるので、それ以上についての推測は控えるべきだとは思いますが、この削除理由を、いまだに「STAP潰し」などと想像されている方もいるようですので、批判を覚悟で書いてみました。
また、「医療維新」(https://www.m3.com/open/iryoIshin/article/204035/)の中に、丹羽氏の説明として
*********
STAP細胞由来のキメラ胚においては、T細胞が、STAP細胞由来か、宿主由来か判定するのが難しい点も述べた上で、「4倍体のキメラでは、『微弱なもの(TCR再構成)が出た』というデータはあった」とした。
**********
とあるのですが、その4Nキメラのデータは公開されているのでしょうか?
ご存じの方がいらしたら、ご教示いただけると幸甚です。
体内時計 さんの『理研は削除した理由を「未発表のデータが含まれていた」と説明したそうですが、釈然としません。』ということですが、矢張り気になりますよね。
先ず、『未発表のデータ』として削除されたものは何か。
—–
○ ES cells
○ Fibrobrast
○ CD45+ cells
○ Sorted Oct4+ #1, 2
× Sorted Oct4+ #3 〜 6
× STAP cluster #1 〜 4
—–
○ CD45+/CD3+ #1(100 ng/50 ng)
× CD45+/CD3+ #2(100 ng/50 ng)
× CD45+ #1, 2
× CD90 STAP #1 〜 3(Sorted Oct4+)
× 2N chimera #1 〜 3(CD45+ STAP)
* ○ が論文に発表したデータの不正検証に用いたレーン。× は未公表データで不正の検証に用いなかったレーン。
「未発表のデータが含まれていた」という理研の削除理由の説明におかしな点は無いとは思います。が、一旦公開したものを削除しなければいけない理由が問題ですね。
まず、Gel1, 2 では全ての TCR 遺伝子再構成のデータでは無いであろうことが推測出来ます。キメラマウスのものが、2N chimera の 3つしかない点、丹羽先生の話によれば、4N chimera のデータもかなり薄いが存在しているということが分かっています。他に未発表のデータがあるということになります。
国際特許出願には“未発表のデータ”のものも公開されていますが、論文のデータが画像解析によって、切り貼りの研究不正が発見されてしまいました。国際特許出願のデータはハーフトーン手法により、画像が劣化していますが、元データと国際特許出願のデータとが第三者に比較検証されてしまい、万が一にも不正が新たに発見されてしまうと、特許の方でも大きな問題になりかねないです。「未発表のデータが含まれていた」というのは事実ですが、特許上の問題回避の為、秘密にしておきたかったのだと思います。
『石井委員会の発表でキメラのレーンが見えないように改訂したのも、突っ込まれて、さらに解析したくなかったからなのかもしれませんね。このときは、理研はなんとか批判をかわしたかったようですからね。』に私も近い印象を持っています。
後々のトラブルは避けたいでしょうから。
誰が「未発表のデータが含まれていた」と言い出したのかですが、それは小保方氏のはずです。
非公開と思われる TCR 遺伝子再構成の図に関しては、共著者である笹井・丹羽両先生が会見等で科学的見解を述べられていますし、公開したあとで非公開にしたものを含めて、今はあまり意味を成さないと思いますが。
oTakeさん
>誰が「未発表のデータが含まれていた」と言い出したのかですが、それは小保方氏のはずです。
ありがとうございました。oTakeさんの仰る通りですね。
先ほど、5ちゃんに以下の書き込みがありました。
「*ttps://www.youtube.com/watch?v=RVp-Rn1ICWM
理化学研究所記者会見~STAP論文調査・最終報告2014年4月1日
1:33:00
中間報告の修正について、小保方さんからの方から未発表データがあるとクレームがあった。」
https://rio2016.5ch.net/life/#2
>「未発表のデータが含まれていた」というのは事実ですが、特許上の問題回避の為、秘密にしておきたかったのだと思います。
なるほど。そういうことだったのですね。深読みしすぎていました。
私は、理研が「STAP細胞由来のキメラマウス」を解析しないはずはないと考えている為、少し想像が膨らんでしまったようです。
>非公開と思われる TCR 遺伝子再構成の図に関しては、共著者である笹井・丹羽両先生が会見等で科学的見解を述べられていますし、
そうなのですね。時間があるときに確認してみたいと思います。
ありがとうございました。
体内時計さん
5chのリンク先は、こちらの方がよろしいかと思います。
https://rio2016.5ch.net/test/read.cgi/life/1537890390/474
軒下管理人さん
ありがとうございました。お手数をおかけして申し訳ありません。
今回の件、軒下管理人さんが仰っていた
「改めて思うのですが、STAPを擁護する方々は、何が面白くてこのような過去の遺物を発掘するのでしょうか?
この特許も、件のブログに投稿されなければ、我々の目に留まることはなかったのに、と思います。
擁護しているつもりで、結果、STAP論文の瑕疵をわざわざ増やして晒しているように感じます。」
に強く同意しています。
小保方氏を一番苦しめているのは擁護の方々なのではないかと、改めて思いました。
軒下管理人さんや体内時計さんのおっしゃる通りだと私も思います。数日前私が筆頭著者のSTAP論文以前の論文の情況をコメントしたら、学様が、今さら解りきっている過去の指摘を持ち出したと怒り狂ってコメントを承認しませんでした。これだってセイヤ氏から「筆頭著者の虚構がSTAP論文以前からと言うなら、それらもES混入だと考えているのか?」という質問があったからなんですよね。筆頭著者の過去の論文がどうなっているかは、STAP論文以前の研究にも虚構があったと考える根拠ですから、書く必要に迫られたのですよね。セイヤ氏の質問が原因で蒸し返されたわけですが、何故かその怒りは私に向けられたのです。このセイヤ氏絡みのエピソードでも解るように、擁護さんたちの主張に反論したり質問に答えるために、筆頭著者の不正行為が延々と蒸し返され続けているのですけど、擁護さんたちは、筆頭著者にとって都合の悪い事実を書かれる=筆頭著者を未だに叩き続けている=批判する方が悪いとしか思っていないのですよね。だからおかしな主張をしては、都合の悪い事実が掘り返されるという無限ループなんです。
https://blogs.yahoo.co.jp/solid_1069/15769264.html#15771140
より、
>特別なミッションを持つ人達の集まりという事がよく解りました。
そんなミッション、ないない。
>もう桂報告書で「けり」がついたはずなのになにが不安なのでしょうか
『デマ』が広がることが不安。
未だに擁護派は桂報告書の結論にケチ付けてるでしょ。ケチ付ける行為が、知らぬ間に『デマ』として広がることを恐れてます。
>擁護派よりとても熱心に小保方Stap否定論陣を続けています。
擁護派が黙れば、必然的にこちらも黙ります。もうオワコンですから。
>不思議です、、、
私も、擁護派の方々に対し、同じ印象です。何故、筆頭著者は研究不正をしていないと、未だに延々と擁護するのか、不思議です。
おしまい。
—–学とみ子氏ブログコメント
学とみ子
あちらのブログでは、以下のコメントありました。
(軒下管理人の“トホホなかーま(笑)。”はどーでもいい話なので省略(^o^))
oTakeさんは以下のように言っています。
>(3) 調査委員会に“小保方氏の TCR 遺伝子再構成の実験結果(PCR)”として提出されたもの(例のGel1 & 2)
(例のGel1 & 2)は、キメラのTCRが出ていることをよくわからない人(小保方氏以外の人が無断で?)が、調査委員会にうっかり提出してしまったのではないですか?
2018/12/2(日) 午後 8:40
—–
『調査委員会に“小保方氏の TCR 遺伝子再構成の実験結果(PCR)”として提出されたもの(例のGel1 & 2)』は、不正調査の対象として、小保方氏自らが調査委員会に提出したものなのですよΣ(-᷅_-᷄๑)
その提出された資料を一般に公開する事自体は、特段の定めがない限り、規定上問題ない。だから、調査委員会は調査資料として Gel1 & 2 の写真図を公開した。ところが小保方氏が『未発表のデータまで公開されている!』と委員会にクレーム付けたわけだ。
キメラマウスの TCR 遺伝子再構成のデータ等は論文に含まれていないのとおかしな話が出てない為、こちらは調査対象外で、そうなると調査委員会も不正調査に使われたデータの部分だけでいいわけで、それで不正調査に使われていない部分をカットして、修正版を出したというわけ。
論文にはキメラマウスを調べたことになっているから、論文に含まなかった理由を笹井・丹羽両先生が会見等で、注入された細胞群に TCR 遺伝子再構成のものが体細胞に分化した際、確率的な問題で検出されない、2N キメラマウスの TCR 遺伝子再構成データはホストのものが含まれているコンタミの可能性、4N キメラマウスのものはかなり薄い等 TCR 遺伝子再構成が見られたとするには問題点があるので論文には含めていないとしたんですよ。
学とみ子氏は状況や話を全然、分かってないでしょΣ(-᷅_-᷄๑)
いや、わざとかね?Σ(-᷅_-᷄๑)
軒下さん。
非常に過度な「正常性バイアス」が即ち擁護派の皆さんの異常性ですから、彼ら彼女ら(順不同)が真相に気がつくはずもありませんね。
この意味では徒労なのですけれども。
「ガベージインガベージアウト」なあのひとたちのタコツボ論壇に光を当てておけば第二波、第三波の擁護派の誕生が難しくなっていくことに寄与することは間違いないと存じます。
でもまあ、アディクションの表明は不味いので、力を抜いてくださいまし。御自愛ください。
なにごとも中庸にやるべきことを淡々となす、これでいきましょう。
偉そうにすみません。
oTakeさん
それ”『調査委員会〜としたんですよ。”を学とみ子さんに言ってほしかったのですよ。
oTakeさんのコメントを読んだ(2018/12/3(月) 午前 7:07 学とみ子:oTakeさん、情報をありがとう)にもかかわらず;
2018/12/3(月) 午前 8:20 学とみ子「調査委員会に、小保方氏が提出したと言うなら、ゲル2の結果について、その時、小保方氏と委員の間でなぜ議論が起きないのでしょうか?つまり、公開してはいけない❗️と。」と、石井委員会に提出したのは小保方氏ではないのではないかと、まだ否定的にとらえています。学様の偏見を改めるのが大変です。ですので、余計ですが、しつこく小保方氏が提出したとコメントしてみました。2018.12.03 09:36 頃。
2018/12/3(月) 午前 9:50 学とみ子 話をそらすコメントがありましたので
2018.12.3 10:21頃 返事をしてみました。
なんかこわれ楽器さんにからまれました。
まず、バッファーくらい自分で作れよとかきました。
あとはやりかたが安田講堂だと。
>学とみ子
何の話しかわかりません。他の方にもわかるようにお願いします。
2018/12/3(月) 午後 10:17
だそうです。ものすごい博士樣がいたもんですね。(゜〇゜;)?????
ハンニバル・フォーチュンさん
お気遣いありがとうございます。
>アディクションの表明は不味いので、力を抜いてくださいまし。
心に留め置きますが、ハードル高いです。努力はしてみましょう。
https://blogs.yahoo.co.jp/solid_1069/15769264.html#15771882
より、
>トホホなかーま(笑)の意味がわかりません。
これを解説しなければならないとは。トホホ。
えーと、このコメントの前の、sighさんのコメントが「トホホ」で結ばれています。そのコメントに同意の意味で「トホホなかーま(笑)。」と私がコメントしました。
お分かりになりましたか?
>トホホ仲間の意味ですか?ため息氏と関係しますか?
前述の通りです。関係していると言えば、関係しています。
>ため息氏が、わざわざ、理研が見せたくなかったキメラのTCRを話題にしてしまったことを言っていますか?
そんなこと欠片も言ってません。
>貴殿からの情報提供を期待しますので、おつきあいをよろしくお願いいたします。
あいにく、提供できる情報は持ち合わせておりません。過剰な期待にはお応えできかねます。
お分かりになりましたか?
大事なことなので、2回繰り返しました。
挙句に”軒先”管理人だって。誰のこと???
おちょくっているつもり??
ったく、救いがたい。
まあまあ。学様の教えの通り相手を好意的に解釈しましょう。とすると、学様はおちょくったり、意識的にとぼけているのではないことになります。すると結論は、ナンチャラ症で記憶を保持できないということにしかならないと思うところです。(笑
学さんのコメント
>何の話しかわかりません。他の方にもわかるようにお願いします。
面白い方ですね。
自分が出鱈目な記載をして、それを指摘された時には、他人に「想像力で読め」という無茶苦茶な逆切れをするのにね。
STAP論文問題について普通の知識と認知能力を有しているなら、何の話なのかは容易に想像できると思いますけどねえ。
「私のなかではでたらめ。バッファーでつみ。」
⇒ STAP論文でバッファーと言えば、小保方氏の記載した実験方法では論文にあるようなpHの培養液は調整できない、という話しかないでしょう。 つまり、この方にとっては、バッファー作成のところで既にこの論文は出鱈目だという判断をさされたということでしょうね。
「一度でた結論に抗うには、答弁ではなくじっしょうで」
「あ、つくるのは酸液とかATP液で結構です。」
⇒ STAP論文は多数の過誤と不正があるため取り下げられた、ということに抗って、擁護派がSTAP論文は正しいと主張するのなら、くだらない屁理屈の答弁ではなく、論文の記載通りにできることを実証すべきである、まずは論文通りのpHの培養液ができるのかどうかを自らやってみるべき、ということでしょうね。
「プロがこうした事をすると、一般人の誤解を煽ります。実際、そうなりました。」
⇒ どんな誤解なのかもわかりませんし、そんな話などなかったはずですが、、 例によって認知機能が正常ではない人の脳内妄想のようですね。
今更ですけど、Gel-2は面白いですよね。レーン27、28の2NキメラのTCR再構成パターンが、CD3+ T細胞とは異なっています。一番下のバンドをプライマーダイマーとして除外すると、一番小さいDJ2のバンドがレーン27、28で見られません。このバンドは一番出やすい(例えば、PCRのかかりが悪いレーン17でも見られます)のですが、2Nキメラでは見られない事から、このキメラでのTCR再構成は、ホスト由来のT細胞のコンタミとは考えにくいです。どのようなサンプルなのか、詳細な情報を出してもらえれば、 科学的な議論が十分可能なデータなんですけどね。
以前にも書きましたが、吉村先生の「せいぜい1本」というのは、過小評価の可能性があります。前提となっている諸条件が、信頼できる考察に基づくのかどうか、よく考えられた方が良いかと思います。
4倍体のキメラであっても、脾臓CD45のSTAPを用いた場合は、B細胞やマクロファージ由来のSTAPからT細胞のレパトアが再構築され、血中を循環すると解釈され、2Nキメラにおけるホスト由来T細胞混入と同様の問題になると思います。なので、微弱な再構成バンドでは、T細胞由来STAPの証明にはならないと思いますよ。
もしデータが公開されているのなら、私も見てみたいですけどね。
Lさん
>4倍体のキメラであっても、脾臓CD45のSTAPを用いた場合は、B細胞やマクロファージ由来のSTAPからT細胞のレパトアが再構築され、血中を循環すると解釈され、2Nキメラにおけるホスト由来T細胞混入と同様の問題になると思います。なので、微弱な再構成バンドでは、T細胞由来STAPの証明にはならないと思いますよ。
ご教授ありがとうございます。
素人考えで、100%キメラなのだから、微弱でもバンドが出ていることはポジティブにとらえていいのではないか、と思っていたのですが、Lさんのわかりやすいご説明でよく理解できました。
ありがとうございました。
一昨年、古田彩氏が、慶應大学で塾生向けに『STAP戦争:謎の”万能細胞”の正体究明に挑んだ研究者たちの300日』というタイトルで講演(http://www2.st.keio.ac.jp/koza/img/poster57b.pdf)されました。
どのようにしてSTAP細胞の正体が解明されていったのか、わかりやすく纏められています。
また、小保方氏が先に論文撤回に同意したのが、不正が認定されたArticleではなくLetterの方だったのは何故だったのか、について、私はもっと単純に考えていましたが、古田さんのご推測が合っているとすれば、残念ながら極めて悪質だと言わざるを得ません。
時間がなく、文章を引用することはできませんが、ご興味のある方は、「慶應義塾大学学術情報リポジトリ」(http://koara.lib.keio.ac.jp/xoonips/)に、「STAP」を入力、検索すると読むことができます。
古田氏は「STAP細胞は本当に作られたのか」と「STAP細胞はあるのか」をいっしょくたにされてしまったのがこの問題の悲劇だと仰っていますが、私もその通りだと思います。
L さん
果たして小保方氏はこのようなデータをシニアの研究者との間で議論してきたのでしょうか?
仮説に都合の良い(誤解でも)と思ったデータしか示さない(実験の天才と褒められたという話がある)とか、若山氏はメチル化のデータを使えないといった(真偽不明)ため、捏造したとの話があるように、シニアとの間でデータをきちんと議論したのか疑っています。
一方、このゲルの記録がきちんとあるのでしょうかね。もはや、なんのサンプルを流したのかわからなくなってしまっているのではないでしょうか。ラベルが本当かどうかも疑わしい。
学生が、予想と異なる結果を持ってくることがしばしばあります。そのときは、しつこく、どういう状況で記録したのかを問いただします。理由が分かる場合もありますし、わからない場合もあります。当方の場合、多くの場合、予想と違うというのはネガティブな結果で、技術的に未熟な場合が多く、仮説をひっくり返すような結果がでてきたことはありませんでした(見逃したのかも)。どうなるかわからない・あるかどうかわからないというような実験もありますが、どちらにしろ、何回も繰り返し再現あるデータを必ず採用しましたけど。
ありがとうございます。舌足らずでごめんなさい。ただ、最後は、プロというかどっかの教授が味方していたので、悲劇のヒロインあつかいする人が増えちゃったという意味です。私のまわりにも結構いました。ま、エア論議は金融だけで。
体内時計さん
古田氏の講演記録ありがとうございます。初めて読みました。遠藤高帆氏の論文妨害のためにLetter を撤回、NGSデータの公開を止めるという推測は説得力がありますね。そうだとするとかなり悪質ですね。ま、それでも公開すべきという意見が通ったので、まともな方もいるということですか。
石井委員会のGel1,2 の生データを一度公開しながら、隠すというのも通じるところがあるように思えます。
山の住人さん アノ姐さん
学とみ子さんに、おちょくる、とぼけるなどの能力を求めてはいけません。狸さんの記事を初めて読んだ学とみ子さんの反応をみれば、明らかですね。
当方もとぼけたことを言うなと批判したこともありますが、真剣だったんですね。真剣にずれているんですよ。
>L さん
『Gel-2は面白いですよね。レーン27、28の2NキメラのTCR再構成パターンが、CD3+ T細胞とは異なっています。』
レーン27と28のラベルを見る限り、2N chimera (CD45 STAP)とありますから、元の細胞 CD 45+(レーン20と21)との比較するのが良いのでしょうけど、ラベル自体、怪しいですから、それは考えないとして、矢張りそれでも PCR では、短いバンドは長いものより効率よく増えるでしょうから…『(一番下のバンドをプライマーダイマーとして除外すると、)一番小さいDJ2のバンドがレーン27、28で見られません。』が気になります…
結局、『どのようなサンプルなのか、詳細な情報を出してもらえれば、 科学的な議論が十分可能なデータなんですけどね。』に尽きるんですが…
使われた試料サンプルがよくわからないですから、科学的に評価しようがない。
小保方氏の作る弱酸性溶液では、論文記載の pH にならない等、最初は塩酸のみで作ったという話であったのに ATP 溶液が殆どで塩酸のものは少しだとか…
論文発表後に丹羽先生が検証実験をされていますが、論文のプロトコル通り(小保方氏の言う通り)弱酸性溶液を作ろうとしたが pH が再現出来ないので、(1) 論文のプロトコル通り(小保方氏の言う通り)弱酸性溶液と(2) プロトコル通りでない、論文記載の pH に合わせた弱酸性溶液(塩酸のみ)の 2 種類を軸に試したが丹羽先生の結果は、『塩酸のみでは(STAP 細胞塊は)作れないことがわかった』というもの。その後、STAP 特許には ATP が使用されている、若山先生が小保方氏付き添いの下で作成したものは ATP が使われていたということなどから、検証実験に於いて、論文外の酸 ATP によるものも検証に追加。
『Callus1 と Callus2 と小保方氏が呼んでいたサンプルを含むが、CD45+ サンプルはなかった。2013 年 3 月の Nature 再投稿に際して、図のスタイル変更とサンプル名付加の要請が小保方氏よりあった。その際、小保方氏は Callus1 と Callus2 がそれぞれ STAP 細胞と CD45+ 細胞であると資料 に書き込み、それに従って図の改訂を行った。(桂報告書p26)』とラベルの変更に関して記載がありますが、これは単なる名前変更ではありません。Callus は、元細胞である CD45+ 細胞等から、酸処理後の細胞に変更がなされています。
この時点で医療事故に於ける 検体取り違え“Specimen mix-up” のような事態になるリスクが高くなり、試料サンプルの信憑性が損なわれます。
仮に情報を出されてもその確かさにかなりの大きな疑問が残ります。
ため息先生の『このゲルの記録がきちんとあるのでしょうかね。もはや、なんのサンプルを流したのかわからなくなってしまっているのではないでしょうか。ラベルが本当かどうかも疑わしい。』に同感です。
『吉村先生の「せいぜい1本」というのは、過小評価の可能性があります。』に関してですが、これはキメラマウスを作成された際の注入細胞群によるバンド数の話だと思います。注入された細胞は十数個の細胞で、その中に TCR 遺伝子再構成が見られる細胞が元々少なく、その細胞が体細胞に寄与したとしても確率的に少ない為、TCR 遺伝子再構成が見られる T 細胞由来のバンドは 1、2 本程度という原理的なロジックです。そこには、キメラマウスのホスト細胞によるものは含まれてない前提ですね。過小評価とは違うのではないかと思います。丹羽先生も同様の説明を会見でされており、実際のコンタミの可能性を考えたデータの解釈の説明になっています。
『STAP 細胞が ES 細胞由来である』との結論がありますが、データの切り貼りによる研究不正、データのラベリングの信憑性、コンタミの可能性、実験に使われた試料サンプルの信憑性等いろいろ考えるとその ES 細胞由来という説明を否定されるような影響が出る話ではないですね。
はい…悪質です。
STAP 事件は、“科学 vs 政治、プロパガンダ”ですね。
私は、遠藤先生の論文公開を妨害するためにletter論文の撤回に先に応じたという古田彩さんの推測から、博士論文の顛末を連想しました。博士論文の疑惑が指摘されたとき、早稲田の教員に論文の取下げを打診したというあのエピソードです。博士論文にしても、NGSデータにしても、これ以上突っつかれては都合が悪い。だから一刻も早く公開を取り止めたかったのではないかしら?でも、letter論文の取下げやNGSデータに関しては筆頭著者と理研の思惑と一致していますよね。理研はletter論文は取り下げられれば調査しないと明言してましたから。筆頭著者も理研もSTAP細胞の正体を隠したかったのだと思いますが、この一致はどう考えたらよいのでしょう?
私は、軒先でも軒下でもどっちでも構わないです。
軒下管理人でエゴサすると、私より数カ月前からお使いの方がいらっしゃるんですね。その方、来年にはそのHNお使いになるのを止めるようですが。ひょっとすると、ご迷惑をお掛けしたのかも。
窓際より厳しい、という位置付けだから、やっぱり私としては『軒下』の方が相応しいなあ。
ため息先生
>遠藤高帆氏の論文妨害のためにLetter を撤回、NGSデータの公開を止めるという推測は説得力がありますね。
科学的知識もあり、取材力もあり、第一線でSTAP問題を見てきた方の意見はそれだけでも十分説得力がありますが、今回のこの推察は目から鱗でした。
古田氏は、丹羽氏に、FI幹細胞の培養条件で培養した場合のES細胞や胚葉体は形態変化について質問されたり、また、伊藤武彦氏にSTAP細胞由来ChIP-seq (input)サンプルについて質問されたりと、会見でも鋭さが際立っていたように思えます。
この講演、おそらくですが、古田氏は、母校の後輩達に研究不正について訴えたかったのではないでしょうか。
須田氏もいつか、早稲田の後輩達に講演をされる日が来るかもしれませんね。若い研究者の卵たちの人生が、研究不正と無関係であることを願わずにはいられません。
風邪は良くなられましたか?
明日の夜から寒くなるそうですので、ご自愛ください。
体内時計さん
お見舞いのお言葉ありがとうございます。単なる風邪だと勝手に診断して、回復しました。
私の場合、よく分かってない学生さんが出してくるデータの方を信じる事が多いです。技術的な問題は、ポジコン、ネガコンを用いた評価を徹底する事によりある程度解決できるので、その上で再現性のある「変なデータ」を持ってくる学生さんとの議論は楽しいです。知恵がついてくると、どういうデータを出すとボスの仮説にあうか忖度したり、論文に載るのはどういうデータか戦略的に考えるようになって、バイアスが入りやすくなります。そこを見抜いて指導していくのは、ちょっとしんどいです 。
小保方さんの場合は、その両方が混在しているように思います。突拍子もないデータもあれば、忖度のデータもあると思います。後者については不正絡みのものも多いだろうと思うのですが、元々は純粋な観察で得られた「変なデータ」から始まった話と思っています。最初から意図的な捏造で作られた話と決めてかかるような論調だと、ちょっとかわいそうかな、と思います(甘いかもしれませんが)。
STAP論文の最大の特徴として、コントロールの欠落が挙げられると私は思っています。コントロールがないと、「変なデータ」の評価はできないですので、この点は、論文投稿される前に指摘されるべきだったと思います。STAP論文の共著者の中で、誰が指導的な立場にあったのかは、議論が分かれるところだと思いますが、 論文作成段階では全著者が「シニア」としての責任を果たすべきだったと考えます。コントロールがないのに、この「変なデータ」をそのまま論文にしてしまった責任は、全著者が負うべきなのに、小保方さん一人に偏りすぎているように感じます。以前に引用されていた本庶先生のTCRコメントでは、笹井先生、若山先生、丹羽先生の責任をかなりきつい口調で指摘されていたと思います。ハーバードや女子医の指導者の責任も大きいと思いますが、何も追及されていませんね。
ゲルに戻ると、Gel-2に関しては、論文に採用されていない2Nキメラサンプルをミスラベルする動機が存在しないように思うので、意図的なミスラベルではないだろうと思っています(エラーの可能性は否定できません)。一方で、特許の方は、十分な動機があると考えており、多少疑いの目で見てます。これらのTCRデータは、複数の「シニア」が見たと思われ、その上で、少なくとも笹井先生と丹羽先生は、コントロール実験の欠落や、システムの不備を理由に、論文から外す決断をしたと思われます。特許あるいはサイエンス投稿バージョンなど、笹井先生、丹羽先生が参加する前の段階では、どのような議論が行われたのか、 情報がないように思います。
あまり議論する意義がないですが、T細胞由来STAPが1個でもあれば、最大で2本のDJ2再構成バンドがでる可能性があります。2個あれば最大で4本です。脾臓のCD45陽性細胞のうち、T細胞は1%から4%とする前提が、間違っている可能性もあります。T細胞STAPのキメラができていたかどうかとは関係ない話です。間違った理論構築に基づいている可能性がある言説を、都合が良いからとそのまま受け入れるのは、擁護の方々の発想と同じであり、あまり感心しません。
L さん
当方の経験からいうと、学生さんの変なデータは技術的な問題の場合が多かったです。原因がわからないときは、再度実験させます。当方ができるだけくっついて。
研究分野が違うので、学生さんが一人ぽっちで実験するということがないから、べったりくっついたりできるわけです。
あら、面白い結果、というのはときどきありますが、なかなか再現しないですね。なんだよ、違う薬物じゃん なんてね。
勿論、学生さんと仮説を建てて、こうなるだろうという予想の元に実験するわけです。学生さんに圧力をかけないように注意するのですが、学生さんにも個性があるから、そんな、教科書的な対応というわけにはいかないときもありますね。難しいです。
Gel2のキメラTCRは、きっと、キメラの体細胞のTCRなんでしょうけど、ホストのT細胞が混ざっているとかのような、あとから考えると実験条件が不十分だったので証拠にならないとシニアの方々が判断したんでしょうね。それが公開されちゃったので、説明が面倒なんで、未公開だから・次の論文につかうから とかにして削除したのではないでしょうか。そもそもSTAP細胞からキメラができないとすると、TCRのバンドはホストの細胞由来しかない。
誤解があるのかもしれませんね。
吉村先生は『実験的にはD2J2のプライマーで検出されるGLをもつT細胞は10%程度ではないかと言われています。もし1個のT細胞がマウスになったとすると、可能性としてはGLもしくは組み換えの1本のみ、GLと組み換えの2本、組み換えの2本、あるいは全く検出できない、となります。よってバンドが見えるとすれば1つか2つ…』という説明をされています。これは注入された細胞の話です。L さんの『T細胞由来STAPが1個でもあれば、最大で2本のDJ2再構成バンドがでる可能性があります。』とほぼ同じだと思います。
これをやり取りされたサイトでは、最終的にホストの細胞が混じってしまっていると判断つかないという話も出ています。
実際の実験の状態があやふやで、ホストのものの混入や複数の不確実な要素が多数あります。笹井・丹羽先生のキメラマウスに対する説明もあるので私はその判断要素となった元図 Gel3 のようなものが他にあるのだろうと推測しています(Gel1 & 2 では数が足りませんし、4N キメラマウスのものも出ていない。)
吉村先生も私もですが、この辺りをはっきりとさせないと最終的に判断出来ないとしています。以前、私の結論は研究不正の可能性もありますが、エラーの可能性もある問題ですというコメントをこちらのブログでしています(確実といえるデータを見ていないのでそういう結論しか出せないので…)
つまり、現状、これ以上は議論にならないということです。
これは余談ですが、笹井・丹羽両先生はこの TCR 遺伝子再構成の実験の詰めが甘かったという会見上でのコメントもあります。
誤解があるのかもしれませんね。
吉村先生は『実験的にはD2J2のプライマーで検出されるGLをもつT細胞は10%程度ではないかと言われています。もし1個のT細胞がマウスになったとすると、可能性としてはGLもしくは組み換えの1本のみ、GLと組み換えの2本、組み換えの2本、あるいは全く検出できない、となります。よってバンドが見えるとすれば1つか2つ…』という説明をされています。これは注入された細胞の話です。L さんの『T細胞由来STAPが1個でもあれば、最大で2本のDJ2再構成バンドがでる可能性があります。』とほぼ同じだと思います。
これをやり取りされたサイトでは、最終的にホストの細胞が混じってしまっていると判断つかないという話も出ています。
実際の実験の状態があやふやで、ホストのものの混入や複数の不確実な要素が多数あります。笹井た・丹羽先生のキメラマウスに対する説明もあるので私はその判断要素となった元図 Gel3 のようなものが他にあるのだろうと推測しています(Gel1 & 2 では数が足りませんし、4N キメラマウスのものも出ていない。)
吉村先生も私もですが、この辺りをはっきりとさせないと最終的に判断出来ないとしています。以前、私の結論は研究不正の可能性もありますが、エラーの可能性もある問題ですというコメントをこちらのブログでしています(確実といえるデータを見ていないのでそういう結論しか出せないので…)
つまり、現状、これ以上は議論にならないということです。
これは余談ですが、笹井・丹羽両先生はこの TCR 遺伝子再構成の実験の詰めが甘かったという会見上でのコメントもあります。
2018/12/18(火) 午後 6:18[ セイヤ ] 君がこの特許の図が論文にならなかったことを笹井氏のコメントで説明してくれました。
笹井氏の説明で問題ないかと思いますが、学とみ子さんは何故答えてくれないのでしょうかね。
それと、セイヤくんは特許の図を使うのは目的外でだめだといっていますが、http://www.soumunomori.com/column/article/atc-160035/を読むと普通の著作物と同じで引用ならいいようですね。
この主旨のコメントを返しておきました。2018.12.18 19:15 頃
セイヤ氏は何かと文句を付けたいんでしょうね。そもそも無断転載とかは、著作権法に基づくもので、論文であろうが、特許公報であろうが、同じです。
学とみ子氏も論文等から図表をブログに転載していますよね?
さぁ、セイヤ氏は学とみ子氏に対し、無断転載だと大騒ぎして下さいな(´-ω-`)
こんなこと言い出したら、この論文関係の図表をブログ等で出した者の殆ど全てといっていいほどアウトになるじゃん(´-ω-`)
アホらし…
oTakeさん
「セイヤ氏は何かと文句を付けたい」ということに同意します。プレスリリースのPTSDから抜け出られず、というか元々かもしれませんが、ケチなことに熱心です。当方の重箱の隅を突っつくことに専念されているようです。趣味なんでしょ。
一方、mjもんた君には成長の証が全くありません。相変わらず「小保方さんの手記や日記に何も反論も出来ないのはどう考えてもおかしい」との念仏ですね。著作権問題における コピペ、引用、転載、盗用、無断 について、それぞれを説明せよ とtrendoブログにコメントしてみました。できないでしょう。
mjもんたtrendo君へのコメントを trendo と学とみ子ブログにコメントしたのでコピー(主旨)を起きます。多分、両方のサイトで承認されないからです。当方のサイトにおいでになる方々は、効果のないことをやってもしょうがないだろとおっしゃるでしょうけどね。2018.12.19 08:20頃。